Biologia dei Tumori e Genomica Funzionale
I progetti in questa area hanno l’obiettivo di investigare gli aspetti dinamici del genoma (quali la trascrizione dei geni e la regolazione della espressione genica, la traduzione e le interazioni proteina-proteina e proteina-DNA) e la loro relazione con I meccanismi che promuovono la formazione e la progressione dei tumori
I principali scopi di questi progetti sono:
- Identificare regioni regolatrici di DNA non codificante coinvolte nei tumori e caratterizzare I loro meccanismi di azione
- Identificare e caratterizzare, sia negli aspetti molecolari che funzionali, long non-coding RNA coinvolti nello sviluppo e progressione dei tumori
- Comprendere la relazione fra genoma codificante e non-codificante nella regolazione della biologia dei tumori
- Esplorare I meccanismi che promuovono l’aggressività tumorale
- Esplorare I meccanismi che promuovono resistenza alle terapie oncologiche
- Studiare I cambiamenti del metabolismo cellulare nella progressione dei tumori.
Prinicipali tecnologie ed approcci sperimentali:
- Chromatin Immunoprecipitation (ChIP) per la caratterizzazione dello stato funzionale del genoma e per l’analisi del legame fra le proteine ed il DNA.
- ChIP-Seq per l’analisi dei profili genome-wide dello stato della cromatina.
- Approcci di genome editing basati sulla tecnologia CRISPR-Cas9, per la mutazione di elementi codificanti e non codificanti del genoma.
- RNA-seq per l’analisi dei profili di espressione genica.
- Luminex-based technology per l’analisi multiplex ad alta sensibilità di analiti circolanti.
- Citoflurimetria a flusso.
- RNA-Immunoprecipitation (RIP-Seq) per l’analisi dell’interazione fra RNA e proteine.
- Chromosome conformation capture (3C) per l’analisi delle interazioni tridimensionali di elementi del genoma.
- Chromatin Isolation by RNA purification (ChiRP) per l’identificazione di regioni del genoma legate da specifici RNA.
- Derivazione di colture primarie da tessuti tumorali e normali.
Lista dei progetti attivi
- Assessing the function of non-coding genome in the metastatic progression of melanom.
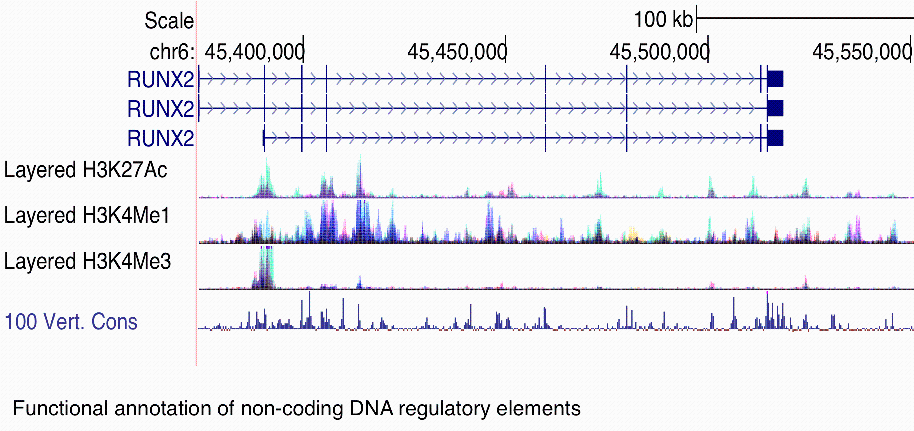
- Characterization of DNA regulatory regions responsible for the aberrant reactivation of Runx2 expression in cancer.
- A CRISPR-Cas9 approach for the identification of genetic determinants of immunotherapy resistance in lung cancer.
- Molecular dissection of Cadherin 6 (CDH6) function in aggressive thyroid tumors.
- From epigenetic drugs to target therapies: improving HDACi based anti-cancer therapies by dissecting the functional relation between HADC and RUNX2.
- RAINs: Novel RUNX2 associated lncRNAs in cancer.
- Novel aspects in the long-non coding RNA (lncRNA) NEAT1 biological function in tumors.
- Characterization of E2F7 role in aggressive thyroid cancer.
- Mapping lncRNAs associated to transcription activator chromatin remodeling complexes.
- Discovery of novel lncRNAs involved in T cell differentiation and Anaplastic Large Cell Lymphomas classification.
- Characterization of SIRT1 pathway in Non-Hodgkin lymphomas.
- SIRT1 regulation in diffuse large B-cell lymphomas: epigenetic modifications as therapeutic targets.
- Characterization of Resveratrol metabolites anti-inflammatory and anti-tumoral potential.
- Investigating Aicda expression and activity in mature B-cell lymphomas.
- Profiling the molecular bases of the metabolitic switch in cancer: a challenge to overcome drug resistance
Prinicipali tecnologie ed approcci sperimentali:
- Chromatin Immunoprecipitation (ChIP) per la caratterizzazione dello stato funzionale del genoma e per l’analisi del legame fra le proteine ed il DNA.
- ChIP-Seq per l’analisi dei profili genome-wide dello stato della cromatina.
- Approcci di genome editing basati sulla tecnologia CRISPR-Cas9, per la mutazione di elementi codificanti e non codificanti del genoma.
- RNA-seq per l’analisi dei profili di espressione genica.
- Luminex-based technology per l’analisi multiplex ad alta sensibilità di analiti circolanti.
- Citoflurimetria a flusso.
- RNA-Immunoprecipitation (RIP-Seq) per l’analisi dell’interazione fra RNA e proteine.
- Chromosome conformation capture (3C) per l’analisi delle interazioni tridimensionali di elementi del genoma.
- Chromatin Isolation by RNA purification (ChiRP) per l’identificazione di regioni del genoma legate da specifici RNA.
- Derivazione di colture primarie da tessuti tumorali e normali.
Lista dei progetti attivi
- Assessing the function of non-coding genome in the metastatic progression of melanom.
- Characterization of DNA regulatory regions responsible for the aberrant reactivation of Runx2 expression in cancer.
- A CRISPR-Cas9 approach for the identification of genetic determinants of immunotherapy resistance in lung cancer.
- Molecular dissection of Cadherin 6 (CDH6) function in aggressive thyroid tumors.
- From epigenetic drugs to target therapies: improving HDACi based anti-cancer therapies by dissecting the functional relation between HADC and RUNX2.
- RAINs: Novel RUNX2 associated lncRNAs in cancer.
- Novel aspects in the long-non coding RNA (lncRNA) NEAT1 biological function in tumors.
- Characterization of E2F7 role in aggressive thyroid cancer.
- Mapping lncRNAs associated to transcription activator chromatin remodeling complexes.
- Discovery of novel lncRNAs involved in T cell differentiation and Anaplastic Large Cell Lymphomas classification.
- Characterization of SIRT1 pathway in Non-Hodgkin lymphomas.
- SIRT1 regulation in diffuse large B-cell lymphomas: epigenetic modifications as therapeutic targets.
- Characterization of Resveratrol metabolites anti-inflammatory and anti-tumoral potential.
- Investigating Aicda expression and activity in mature B-cell lymphomas.
- Profiling the molecular bases of the metabolitic switch in cancer: a challenge to overcome drug resistance
Ultimo aggiornamento: 02/10/23